رویکرد جدید به تحقیقات پروتئین با هوش مصنوعی
در مقالهای که به تازگی در مجله Nature Communications منتشر شده است، تیمی از محققان رویکردی مبتنی بر هوش مصنوعی را برای بررسی شباهتها و روابط ساختاری در دنیای پروتئینها ارائه کردهاند. این تیم شامل اعضایی از دانشگاه ویرجینیا است، از جمله فیلیپ بورن، رئیس دانشکده علوم داده، کام مورا، دانشمند ارشد این دانشکده، و الی درایزن، یکی از فارغالتحصیلان اخیر این دانشگاه.
مطالعه آنها دیدگاههای سنتی درباره روابط ساختاری پروتئینها (یعنی الگوهای شباهتها و تفاوتها) را به چالش میکشد و در این فرآیند، بسیاری از روابط ضعیفی را که توسط روشهای سنتی نادیده گرفته میشوند، شناسایی میکند.
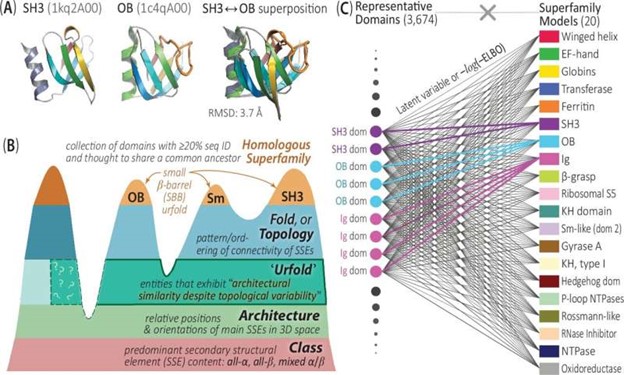
نویسندگان این مطالعه به طور خاص یک چارچوب محاسباتی را گزارش میدهند که قادر است این روابط پروتئینی را در مقیاس وسیع (در میان پروتئینهای متعدد) بهطور دقیق شناسایی و کمّی کند. این چارچوب به روشی جدید، منعطف و با جزئیات زیاد، با استفاده از روشهای یادگیری عمیق و یک مدل مفهومی جدید به نام “Urfold”، این امکان را فراهم میآورد که دو پروتئین با وجود تفاوت در تاخوردگیها یا “فولدها”، شباهت معماری داشته باشند.
بورن، مورا و درایزن در این پروژه با استلا ورتنیک همکاری کردند. همه نویسندگان از اعضای آزمایشگاه علوم زیستی محاسباتی بورن و مورا هستند که بخشی از دانشکده علوم داده و بخش مهندسی زیستپزشکی دانشگاه ویرجینیا است.
این مقاله نتیجه سالها تلاش آزمایشگاه بورن برای توسعه یک چارچوب مبتنی بر هوش مصنوعی به نام DeepUrfold است که به منظور بررسی سیستماتیک و گسترده نظریه Urfold در مورد روابط ساختاری طراحی شده است.
با استفاده از DeepUrfold، تیم آزمایشگاه بورن توانست روابط ساختاری ضعیف بین پروتئینهایی را در سراسر جهان پروتئینها شناسایی کند که پیش از این از نظر تکاملی یا به هر دلیل دیگری، غیرمرتبط تلقی میشدند.
DeepUrfold در شناسایی و توصیف این روابط دوردست، پروتئینها را بهصورت “اجتماعات” در نظر میگیرد و از رویکرد سنتی دستهبندی پروتئینها در بخشهای جداگانه و غیرهمپوشان اجتناب میکند. این روشهای جدید میتوانند پژوهشگران را ترغیب کنند تا از تفکر ایستا و هندسی درباره شباهتهای پروتئینی فراتر روند و به سمت یک رویکرد یکپارچهتر حرکت کنند.
بورن، که بنیانگذار و رئیس دانشکده علوم داده است، در جامعه علمی به دلیل تحقیقاتش، از جمله در زمینه بیوانفورماتیک ساختاری و زیستشناسی محاسباتی بهطور کلی، شناخته شده است. او در اوایل دوران حرفهای خود، در توسعه بانک داده پروتئین RCSB همکاری داشت؛ این منبع ارزشمند از اطلاعات ساختار پروتئینی به انقلاب در این حوزه کمک کرد و راه را برای پیشرفتهای هوش مصنوعی مدرن مانند AlphaFold هموار ساخت.
مورا که در دانشکده علوم داده و بخش مهندسی زیستپزشکی دانشگاه ویرجینیا فعالیت میکند، در زمینه زیستشناسی ساختاری و محاسباتی، از جمله مطالعات بیوشیمیایی و کریستالوگرافی سیستمهای مبتنی بر RNA و بیوفیزیک مولکولی DNA، تخصص دارد. او به سیستمهای زیستی از دریچه تکامل مولکولی نگاه میکند و به تقاطع این حوزهها با علم داده علاقهمند است.
درایزن، که دکترای مهندسی زیستپزشکی خود را زیر نظر بورن از دانشگاه ویرجینیا دریافت کرده، هماکنون به عنوان پژوهشگر پسادکترا در زیستشناسی محاسباتی در دانشگاه کالیفرنیا، سانفرانسیسکو مشغول به کار است. ورتنیک نیز محقق ارشد دانشگاه ویرجینیا است که تمرکز او بر زیستشناسی محاسباتی، ساختار، عملکرد و تکامل پروتئینها است.
گفتنی است علاوه بر نوبل فیزیک، برندگان نوبل شیمی امسال، به دلیل توسعه روشهای پردازشی با کمک هوش مصنوعی برای پیشبینی ساختار پروتئینها به این جایزه دست یافتهاند. باید دید توسعه روشهای پردازشی پیشرفته با کمک هوش مصنوعی، علاوه بر داروهای جدید، چه دستاوردهای پیچیده و جذابی را به همراه خواهد داشت؟
منبع: Phys.org
مقاله مرتبط: https://dx.doi.org/10.1038/s41467-024-52020-2




